SCIENCE | 空间组学的曙光

空间组学已被广泛誉为生命科学的新前沿。这一术语涵盖了广泛的技术,这些技术有望改变生物学的许多领域,并最终通过同时测量物理组织结构和分子特征来彻底改变病理学。虽然该领域在过去5年中发展成熟,但仍存在一些成长的痛苦:进入障碍、稳健性、最佳实验设计和分析实践不明确、缺乏标准化。在这篇综述中,作者对不同的空间组学技术家族进行了系统的分类;强调他们的原则、力量和局限性;并就这一令人难以置信的强大但仍然难以驾驭的景观中面临的最大挑战给出一些观点和建议。
生物学的复杂性主要是在三维(3D)领域中展开的。虽然我们经常研究蛋白质的空间结构,但要深入研究更简单的生物体或个体组织,则需要分析无数细胞的分子谱,并了解它们的空间排列如何影响它们的行为。近年来,多路空间分子测量技术的创新开启了生物研究的变革时代,使人们对生命系统的复杂性有了更深入的了解。
这一转变的起源可以追溯到应用于分解组织的单细胞“组学”技术的出现。这些工具促进了新型细胞类型的鉴定,为生物进化提供了新的见解,并启动了人类和小鼠组织的全面数据库。关键是要认识到组织中细胞的空间定位深刻影响它们的功能。例如,一个细胞的位置决定了它与其他细胞的相互作用,主要是通过接触或局部信号传导,从而形成像各种组织干细胞那样的专门环境。物理和化学属性的空间变化在调节生物活动中起着至关重要的作用,特别是在多细胞生物发育过程中。然而,对分解细胞的标准测量遗漏了这一空间维度,这推动了“空间组学”(spatial omics)的发展——旨在在其3D环境中绘制细胞的分子方面的技术。
空间组学技术的主要视角是蛋白质及其改变或mrna,但他们现在也在探索空间基因组和表观基因组方面。与传统技术相比,空间组学提供了更表层的分析,经常在实验时间的基础上权衡空间精度。尽管如此,这一新兴领域已经取得了显著突破,揭示了与患者结局相关的动物进化、大脑组成和肿瘤微环境等领域的见解。我们只触及了空间组学范式的表面,但它的快速发展预示着对生物学的更丰富、情境驱动的理解。
The current generation of spatial molecular profiling tools
虽然对组织切片进行空间分子评估的概念由来已久,免疫组织化学(IHC)和原位杂交(ISH)等工具已经使用了50多年,但它们的复用能力受到可区分染料数量的限制。为了达到“组学”级别的精度,简单地添加更多的染料是不可行的。因此,空间组学技术转向替代技术。这些方法包括非显微方法,如质谱(MS),涉及迭代染色和去除染料以实现所需复杂性的程序,或者利用高通量DNA测序联合条形码的效率。
Multiplexed antibody profiling
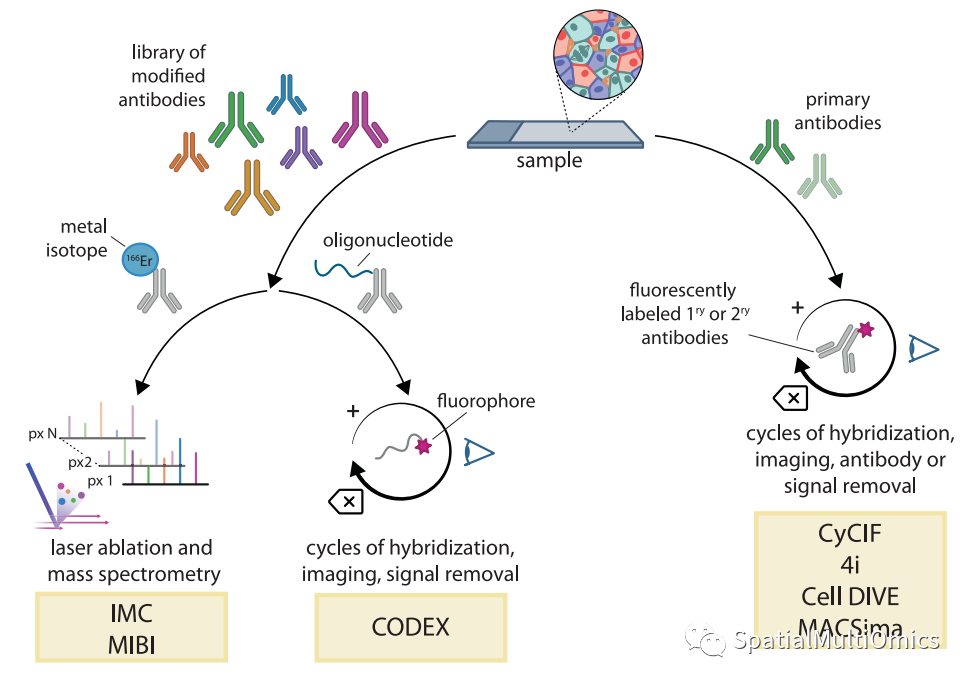
在过去的20年里,质谱(MS)已经被有效地用于原位评估。新一代被称为“质谱细胞术”(MC),依赖于与同位素纯镧系金属相关的抗体检测。每个抗体通过螯合聚合物连接到一种独特的金属上。在成像过程中,利用激光逐像素汽化组织。由于生物材料中没有镧系元素,而且能够精确区分它们的质量,因此可以对40多种具有特殊信噪比的实体进行量化。两种空间成像技术,"成像质谱细胞术" (IMC)和"多路离子束成像" (MIBI)是基于这一原理建立的。最初设计用于通过抗体结合检测蛋白质,现在已经通过金属连接的杂交探针进行了RNA成像。然而,由于可分辨物质的数量有限和金属的纯度,复杂性限制在大约50种。然而,空间MC方法取得了巨大成功,特别是随着工具和试剂的商业化。它们的应用涵盖了数百项研究,其中免疫学和癌症生物学等领域尤其受益。例如,一项对约500例人类乳腺癌样本进行的研究显示,特定的肿瘤微环境(TME)模式与患者结局密切相关。
另一种方法是在重复成像周期中使用常规标记的荧光抗体。在一些技术中,所有的抗体都是同时连接并通过二次探针进行循环检测,如“通过索引进行共同检测”(CODEX)。在其他方法中,抗体是循环结合的,要么在每个检测阶段后被去除,要么使用光漂白等技术使其不可检测。这些方法,包括CyCIF, Leica Cell DIVE和Miltenyi MACSima,各有其优缺点,特别是在周期速度和抗体微调方面。但总的来说,他们在数据生产上超过了微软。循环成像技术是处理大量样本集的项目的最佳选择。
Spatial transcriptomics and genomics
“空间转录组学”技术的目标是在整个组织样本的亚细胞水平上以3D的方式测量每个基因及其变体的存在。然而,目前没有任何工具能够完全实现这一点。所有技术都在灵敏度、分辨率和用户友好性等方面做出了妥协。目前,这些技术大多集中在组织学切片,基本上在2D功能,尽管也有例外。
空间基因组分析的基础是基于四个主要策略:
Microdissection: :这涉及物理分离或标记特定组织区域,使其DNA或RNA的提取和分析成为可能。
Combinatorial fluorescence in situ hybridization (FISH):通过FISH后的单分子成像,该方法可以对特定组织切片中的mRNA分子进行计数。
In situ sequencing: 对组织内的mRNA分子进行直接测序。
Spatial barcoding: 在这里,来自特定区域的DNA和mRNA被连接到一个独特的DNA“条形码”。批量测序后,这种条形码有助于将它们映射回它们在组织中的原始空间位置。每一种策略都有其细微之处和细节。
Microdissection and photo-isolation
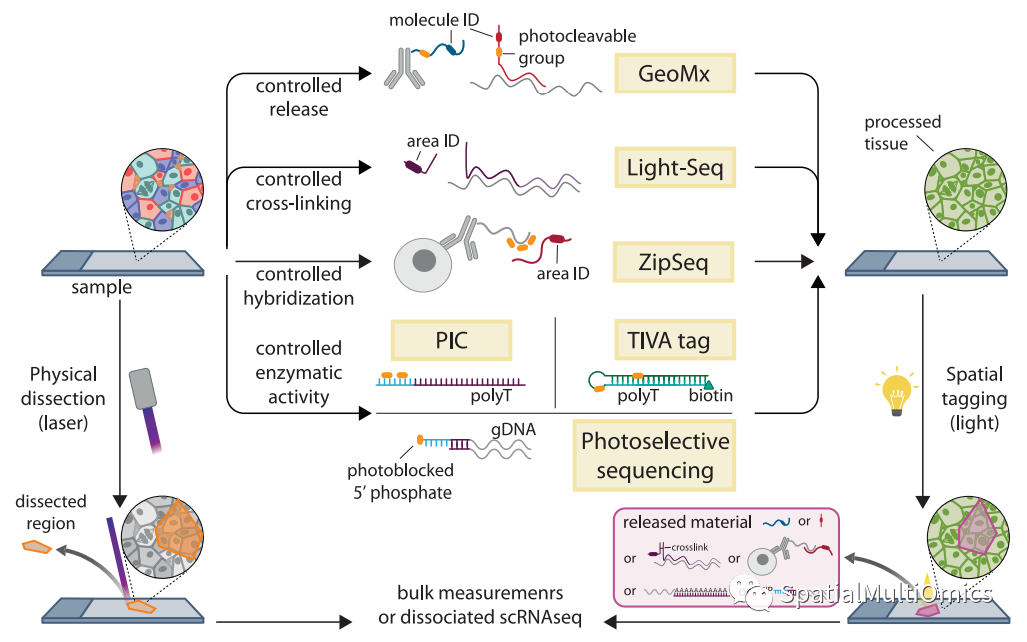
显微解剖是一种通过物理分离从特定区域提取生物分子,从而为分子图谱添加空间背景的方法。激光捕获显微切割(Laser-capture microdissection, LCM)是一种经常用于分离小块(从数十个到数百个细胞)进行rna测序(RNA-seq)的技术,它可以实现单细胞精度。
创新已经将光作为一种工具,取代了物理分离来确定要对哪些区域进行分析。光在这方面有几个应用:
触发逆转录(RT):转录组在体分析(TIVA)标签和光隔离化学(PIC)等技术利用了这一原理。
激活报告基因:Nanostring GeoMx方法证明。
附加纯化标签:如Syncell MicroScoop技术所见。
清除障碍:如在选择性光测序(PSS)中,阻止测序适配器连接的障碍。
启动DNA条形码的连接:光可以通过促进DNA条形码的交联或引导其与组织杂交来催化DNA条形码与特定区域的连接。
所有这些技术,无论其方法如何不同,都有共同的优缺点。它们的优势在于能够提供类似于批量测序的深度分析,以及灵活调整研究领域(从单个细胞到广阔区域)的能力。另一方面,它们的吞吐量通常很低,大多数低于数百个位置,因为每个选择的区域需要单独的收集和处理。尽管条形码技术在一定程度上缓解了这一限制,但它们最多也仅限于数百个领域。
Multiplexed ISH
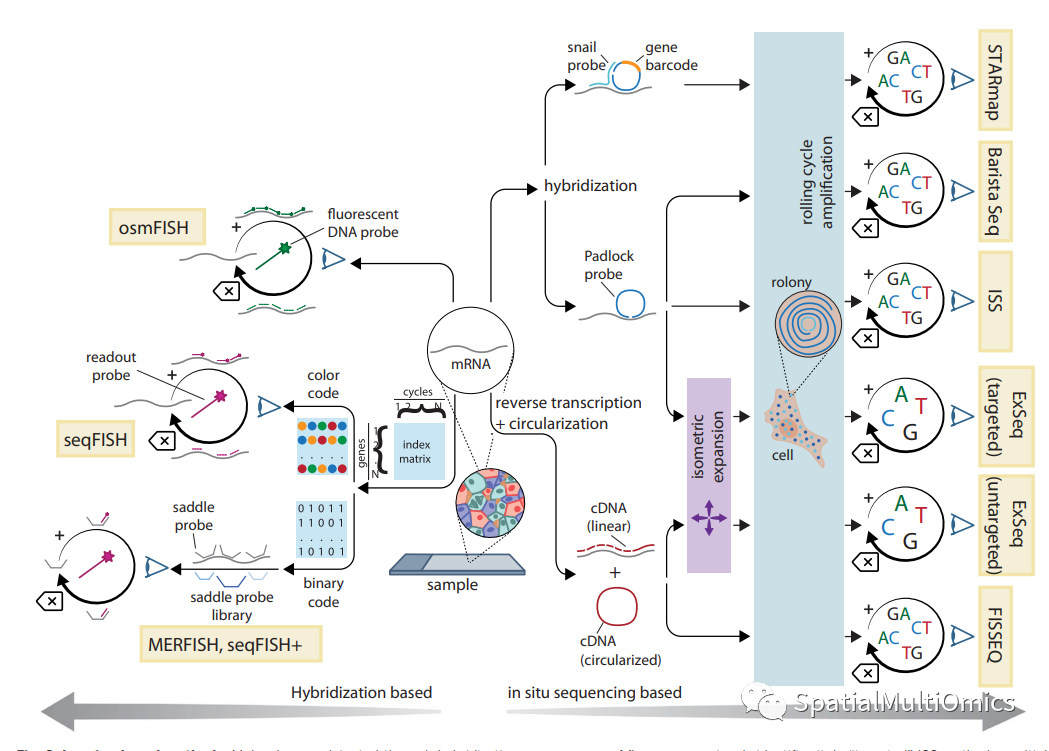
基于循环杂交的方法起源于单分子FISH (smFISH),将组织中单个mRNA分子定位为微小的荧光点。为了产生可检测的信号,几个探针必须附着在一个mRNA分子上,要么是因为许多荧光团结合在一起产生信号,要么是因为两个紧密结合的探针触发信号放大。smFISH以其精确的RNA定量被认为是"金标准"。因此,由其产生的空间分析技术通常表现出卓越的敏感性。
所有循环FISH技术都有以下共同特点:
通过DNA探针杂交将组合条形码与转录本联系起来。
利用荧光成像循环读取条形码。
通过多个探针或信号放大在每个分子上产生可检测的信号。
重置信号后循环。
序贯荧光原位杂交(seqFISH)采用荧光素标记的FISH探针,直接与细胞的mrna结合。在每个成像周期后,使用脱氧核糖核酸酶(DNase)消化去除它们。相比之下,多路鲁棒误差FISH (MERFISH)使用带有mrna结合片段和“报告”部分的探针,用于条形码元件,并通过后续杂交检测。这种检测方法实际上不依赖于rna,增强了对核糖核酸酶污染的抵抗力,缩短了成像时间。
近年来,MERFISH和seqFISH+已经升级为全转录组水平的应用,扩展到mRNA和蛋白质的联合检测,并通过信号放大方法进行改进。merfish -扩增显微镜融合可以识别在其他情况下难以区分的近端转录本。MERFISH和seqFISH+在敏感性和通量方面产生了相似的结局,但有细微的方案差异。虽然MERFISH现在已经商业化,而且更容易使用,但其复杂的技术性质阻碍了其广泛使用。
除了MERFISH和seqFISH之外,还有几种技术具有类似的特性。衔尾蛇smFISH (osmFISH)缺乏条形码,每个周期检测的转录本有限,但方案简单。Saber-FISH和Clamp-FISH强调信号放大,提高了信噪比(SNR)。在福尔马林固定的石蜡包埋组织中,SCRINSHOT靶向约30个转录本。增强的电FISH (EEL-FISH)利用电泳将细胞mrna推到导电载玻片上,然后去除组织,从而获得更高的信噪比和速度。在商业上,几种可同时分析数千个基因和多种蛋白质的选择正在出现。
In situ sequencing
原位测序技术与循环fish方案非常相似。他们直接在组织学切片中进行高通量测序化学,最常用的技术是连接测序。所有这些方案通常都是通过从检测到的转录本衍生的模板滚环扩增(RCA)来创建DNA“纳米球”或克隆。每个mRNA分子产生一个单独的rony,促进转录本计数。与传统的流式细胞测序相比,由于原位操作的复杂性,原位测序被限制在大约30个核苷酸的读长。
ISS方法是一种早期的迭代,采用了与mRNA结合的挂锁DNA探针。这些探针要么确保它们的末端在连接时完全重叠,要么在连接前形成一个由聚合酶填补的间隙。根据变异的不同,测序可能针对条形码序列或所创建的间隙中存在的任何序列。结果是基因表达谱分析(计数与预定基因集的杂交)或特定基因列表的靶向从头测序。随着时间的推移,该方法不断发展,选择了杂交测序,这有效地成为了在条形码上执行的seqFISH-MERFISH的一种专门形式,产生了更好的信噪比。
另一种方法是荧光原位测序(fluorescence in situ sequencing, FISSEQ),通过使mrna逆转录产生的cDNA分子环状产生RCA扩增子。这实现了真正的空间测序,但由于原位RT和cDNA循环效率低,读取长度有限,灵敏度低。
随后的方法,如STARmap和BaristaSeq,从ISS和FISSEQ演变而来,提高了效率并加入了新的功能。例如,STARmap使用两个探针来提高灵敏度,而BaristaSeq则改进了间隙填充挂锁探针方法。最近,基于挂锁的ISS和FISSEQ与扩增显微镜化学技术相结合,提高了效率和信噪比。一种名为ExSeq的创造性方法将短读长ISS与典型的“批量”测序融合在一起,使精确的长读长ISS能够识别复杂组织(如小鼠大脑)中的剪接亚型。
fish和原位测序方法的共同之处是最后的检测步骤,这涉及组织的高分辨率显微镜成像,通过有效计数单个mRNA分子来测定基因表达。单分子显微镜的挑战在于其仪器和操作的精度要求很高。图像的共配准和特征提取是一个突出的挑战,因为有限的视场是缓慢的过程。此外,由于衍射极限,细胞内的分子拥挤设置了分辨率边界,使检测灵敏度偏向于更大的细胞,从而允许更深入的分析。
Spatial barcoding of bulk sequencing reads
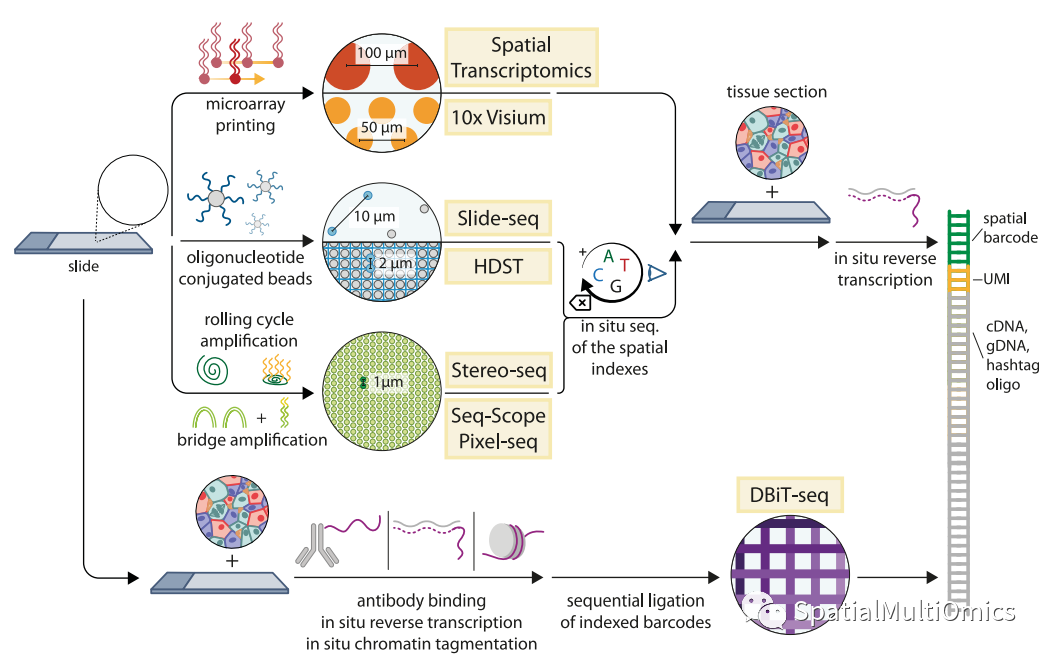
二代测序的潜力在于能够快速产生高质量的转录组数据。如果空间定位可以编码,它将是无价的。这是空间转录组学(ST)背后的概念。在这个过程中:
利用微阵列印刷将寡核苷酸序列排列在玻片上。
每个寡核苷酸都包含一个测序兼容的手柄、一个空间特异性条形码、一个调整PCR偏倚的唯一分子标识符(UMI)和一个捕获多腺苷化信使的聚胸苷序列。
每个阵列点上都有不同的空间条形码。
阵列上设置组织学切片,使细胞RNA能够连接到条形码寡核苷酸,并在原位进行逆转录。这将生成空间索引的cdna。
cdna被扩增,转化成文库,然后通过标准二代测序进行测序。
ST的优势包括:
产生较长的测序片段,尽管存在3 '端偏倚。
不依赖复杂的成像设备。
快速处理,不依赖于样本大小。
适合并行化。
这些好处推动了它的商业接受度。然而,ST存在分辨率仅为50 ~ 100 μm、样本成本高、RNA捕获效率欠佳等缺点。
为了提高ST的分辨率,随后开发了两种方法:Slide-seq和高密度ST (HDST)。这些方法:
将条形码poly(T)引物附着在纳米珠上,而不是玻璃表面。
实现更好的空间密度,Slide-seq分辨率为10 μm, HDST分辨率为2 μm。
改进版Slide-seq V2和另一种方法Slide-tags分别利用这一策略提高了RNA捕获效率和多组学空间分析。
Seq-Scope是一种利用广泛采用的下一代测序平台的创新方法。它:
使用改良的测序流式细胞仪,通过桥接PCR扩增产生DNA条形码。
实现一个密集的条形码幻灯片,不同的空间条形码大约每500纳米。
另外两种技术,Stereo-seq和PIXEL-seq,使用载玻片上的DNA纳米球来获得亚微米的分辨率,具有值得称道的RNA捕获效率。
另一种方法,DBIT-seq,使用组织上的微流体通道来创建物理屏障。条形码寡核苷酸流经通道并与靶分子结合,形成独特指数网格。这种方法可以单独或联合分析RNA、蛋白质和染色质特征。这种捕获效率是值得称赞的,主要是因为不需要rna扩散到条形码上。
虽然空间条形码结合批量测序由于商业化、易用性和与现有工作流程的兼容性而取得了成功,但挑战仍然存在。空间条形码的低分辨率和任意定位可以使不同细胞类型的结果平均。然而,ST和相关技术已经在神经科学和癌症生物学中得到了有益的应用,随着技术的进步,ST和相关技术可能进一步促进其在空间分析中的应用。
The balancing act of spatial profiling
在最佳环境下,空间分析技术将快速、可靠、经济地提供细胞的全面、高分辨率分子信息。然而,目前的技术涉及权衡取舍,因此方法的选择取决于研究的要求。需要考虑的关键因素包括分辨率、吞吐量、易用性、健壮性和成本。
蛋白质分析
IMC-MIBI与循环免疫荧光:
IMC和MIBI:它们都是商业化的,坚固的,并且被广泛使用(尤其是IMC)。利用该技术的预偶联试剂种类越来越多,也有许多成功的研究。局限性包括标记物少于50种、灵敏度有限、成本高、成像时间长和分辨率相对粗糙。MIBI提供了更好的分辨率,但速度较慢。
循环免疫荧光:更实惠,更高的通量,它可以检测超过100个特征,有足够的时间和抗体可用。该技术具有高分辨率,可用于天然抗体。但其信噪比(signal-to-noise ratio, SNR)低于MS,且多次循环后会对组织造成损伤。
空间转录组
基于成像的方法:对于高分辨率,例如识别不同细胞区室中的转录本,这些方法更优越。但是,由于需要大量的平铺和图像处理,它们是耗时的。
杂交:更敏感,但往往产生较弱的信号。最适合较薄的组织切片。
原位测序:使用信号放大,适用于各种组织类型,并能处理较厚的样本。但灵敏度较低,信号放大可能进一步降低灵敏度。
非靶向分析:原位测序方法(如ExSeq)允许在不预先定义探针的情况下进行全转录组分析,从而有助于了解剪接异构体和突变。然而,由于原位放疗效率低,其灵敏度往往低于靶向方法。
Barcoding-based方法:
空间转录组学(ST):这种流行的方法每个条形码的分辨率为20-100个细胞,使得详细的空间分析具有挑战性。
其他协议:它们提供从10 μm(幻灯片-seq)到~500 nm(如PIXEL-seq和Seq-Scope)的分辨率。DBIT-seq,一种微流控方法,达到了10 μm的分辨率。这些方法都不是“cell-aware”的。
优点:它们在吞吐量方面是有益的,因为采集时间不随样本大小或特性数量的变化而变化。多个样品可同时处理。
激光捕获和空间标记(例如TIVA, PIC):这些方法是深入分析特定空间位置的理想方法。它们相对简单,允许直接提取物质进行批量分析。
从本质上讲,虽然空间分析技术取得了长足的进步,但每种技术都有其优势和局限性。技术的选择在很大程度上取决于研究的目标和限制。
Big data to useful data—the challenge of analysis
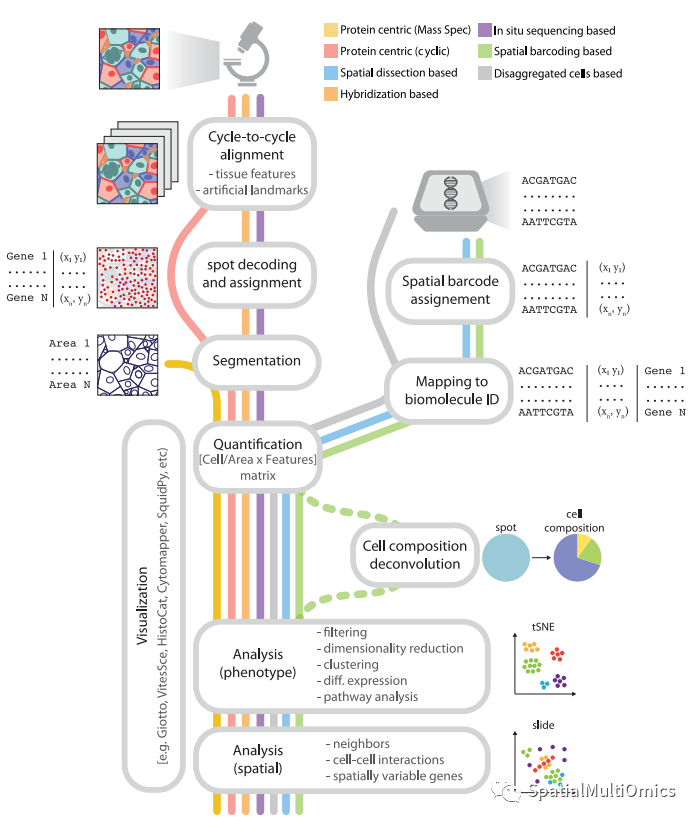
空间组学方法不仅仅是数据获取,数据操作、分析和可视化也同样重要。这些方法产生大量的数据集(有时是tb级的),如果没有适当的分析工具,这些昂贵的实验可能会变得无用。此外,由于数据的巨大规模,需要专门的硬件和软件来进行可视化。
分析管道:
基于图像的方法:最初的挑战是重新排列不同周期的图像并将其拼接在一起。图像配准,即对多个图像进行相互对齐,由于数据量巨大,可能会非常复杂。大多数方法使用“基准标志”或荧光珠使注册更简单,但它需要额外的实验处理。
转录组学方法:这些方法需要解码荧光信号序列与特定基因匹配或被解析为新序列。在此步骤中应用错误校正和检测。
加工流程:每一种技术或显微镜都需要修改,因此很难标准化。尽管如此,SpaceTx联盟最近推出了一套多功能的Python模块Starfish,尽管专门的管道常常优于它。
分割和数据处理:
图像分割:这个过程识别原始图像中属于单个细胞的特定区域。这得益于人工智能和深度学习的进步。细胞核的分割由于其规则的形状是直接的,确定细胞质膜的边界是比较棘手的。开发一种通用的分割膜标记将彻底改变空间组学领域。
空间条形码方法:由于数据类似于传统的测序方法,这些方法有一个优势,因为它们绕过了许多图像分析。然而,这些方法不是将数据映射到特定的单元格,而是映射到不同大小的“箱”。对于高分辨率方法,可以合并多个bin来创建单元级数据库,重新引入图像分析的需求。
后处理:完成上述步骤后,数据集格式与分解的单格分析方法相当,但每个格都有空间坐标。
数据分析和可视化:
可以使用Seurat、ScateR、Scanpy和Monocle等工具,但由于可能存在偏差,必须谨慎使用。一些用于空间数据的工具已经升级,同时也开发了专用的软件包。
传统的降维和聚类工具可以处理空间数据,但不能充分利用其潜力。然而,有效利用空间数据的工具的开发正在增长,重点是:
识别空间可变标记和特定表达模式。
用一组共表达标记识别空间龛。
检测细胞类型或邻近组织之间的空间相互作用。
随着空间组学方法的快速发展,该领域需要开发统计框架和分析工具来利用大量数据。
The challenge of experimental design
空间分析技术是一种新颖的工具,在众多的应用中有着巨大的前景,但明智地使用它们至关重要,重点关注那些真正需要空间组学的问题。这些技术的一个关键用途是在各种条件下识别组织中的新细胞类型和状态,这是人类细胞图谱计划的目标。空间组学可以突出传统方法可能忽略的基因或蛋白质表达差异,并将这些差异与空间定位联系起来。他们可以研究不同细胞类型的共定位如何影响它们的特征,这在研究肿瘤进化和对治疗的反应方面有应用。这些技术能够提供组织在自然状态下的详细信息,使深入的原位检测成为可能。
这些工具可以识别单元格类型和状态,甚至在考虑空间细节之前。根据组织的空间一致性,相似类型的细胞可以根据已知的组织解剖结构或利用空间信息来定义“邻近簇”或分数。空间组学的一个重大挑战是每个位置测量的特征数量有限。为了减轻这种情况,空间测量可以与分解的单细胞方法相结合,这些方法可以提供更广泛的分析。集成这些数据集需要重叠的标记,并且只有当两种方法都能捕获样本的完整细胞类型和状态异质性时才有可能。
复制是一个核心问题,特别是对于像肿瘤这样具有不同空间结构的组织。随着数据分辨率的提高,即使看似具有一致结构的组织也可能显示出样本间的差异。这对于定义细胞类型或状态之间的边界可能是有问题的。关于复制数量的建议没有固化。虽然许多研究依赖于至少三次重复,但更稳健的方法包括初步的预试验来测量方差,然后与统计学家合作来确定理想的样本量。但是,财务考虑和技术限制可能会影响可行的重复次数。在研究罕见现象时,获取足够的样本至关重要。使用全玻片扫描或3D全器官显微镜等技术对组织进行预筛选,可以精确定位进行详细分子分析的区域。最优空间组学研究往往结合多种互补技术。
Future directions
空间组学时代刚刚开始,近年来的快速发展预示着更多的革命即将到来。
空间分析方法预计将变得更快、更高效、更强大。这些方法的商业化正在扩大,有几家公司已经提供了交钥匙解决方案,还有更多公司将紧随其后。这种商业增长将导致更广泛的可及性,促进空间组学的民主化。
有一个明显的趋势,那就是画像深度的增加。基于影像学的RNA测定技术已经在细胞培养物中达到了全转录组水平,这一技术可能在组织中得到复制。对于蛋白质,虽然全蛋白质组检测仍然难以实现,但我们可以预期可测量标志物的数量将显著增加,这可能需要放弃基于ms的方法和创建特异性抗体。然而,由于实验的复杂性增加,对更深入测量的推动可能仍然局限于小众应用。我们期待非靶向原位测序和空间条形码的特异性和分辨率会有所提高,而后者可能成为大规模研究的首选。
还可以预见将技术扩展到更广泛的测量范围。虽然大多数方法关注的是mRNA丰度或蛋白表达,但最近的技术现在允许使用多重免疫染色方法进行RNA定量。另一方面,寡核苷酸偶联抗体可用于循环杂交和空间条形码技术。此外,基因组和表观基因组测量越来越多地在空间上执行。seqFISH+、MERFISH和空间条形码方法等技术集成了基因组和表观基因组分析。应用ISS技术检测DNA点突变是可行的。
虽然这些分析模式主要是一次执行一两个,但未来空间多组学技术有望激增,这反映了在分解的单细胞工具中看到的进化。我们的最终目标是开发出整合基因组学、转录组学和蛋白质组学-小分子检测的技术。
人们预计会转向真正的3D轮廓。目前,大多数空间组学是二维的,在薄的组织切片上操作。这对处理厚样本的基于图像的分析和条形码方法提出了挑战。目前的2D方法缺乏对组织的全面视图,只有某些复合IHC和ISS方法近似于3D轮廓。一种促进3D轮廓的解决方案是通过2D技术处理3D样本的连续薄层,然后通过计算将数据对齐到3D模型中。这种方法虽然耗时,但已经在各种平台上得到了展示。新的3D分析策略可能会在未来几年出现。
不久以前,空间组学的能力还被认为是科幻小说。今天,这些技术正在迅速发展,为研究人员提供了对复杂生物系统无与伦比的见解。随着这些进步,在理解这些工具的局限性和最佳实践方面也面临着挑战。随着这些尖端技术的普及,突破性的生物学发现是不可避免的。
免责声明:本文旨在传递更多科研资讯及分享,所有其他媒、网来源均注明出处,如涉及版权问题,请作者第一时间联系我们,我们将协调进行处理,最终解释权归旭为光电所有。



![[文献速递Vol.215]-SNR-Net OCT:通过深度学习对低光光学相干断层扫描图像进行增亮和去噪](https://www.surisetech.com/wp-content/uploads/2024/01/wen-xian-su-di-vol215snrnetoct-tong-guo-shen-du.png)