纳米分辨率光学显微成像
所谓眼见为实,人类通过感官从自然界获取的各种信息中,以视觉信息占比最高,约占获取信息总量的80%。然而人眼的分辨率极限为100 μm左右,大概是一根头发的直径,因此仅能对宏观尺度生物个体及器官、组织进行观察,但无法直接观察细胞、亚细胞等微观世界的生命活动。17世纪,列文虎克和罗伯特·胡克对光学显微镜的创造性贡献及应用,为人类提供了探索生命活动微观世界大门的钥匙,光学显微镜具备对微米尺度的细菌、细胞及其内部结构进行观察的能力(图1)[1]。
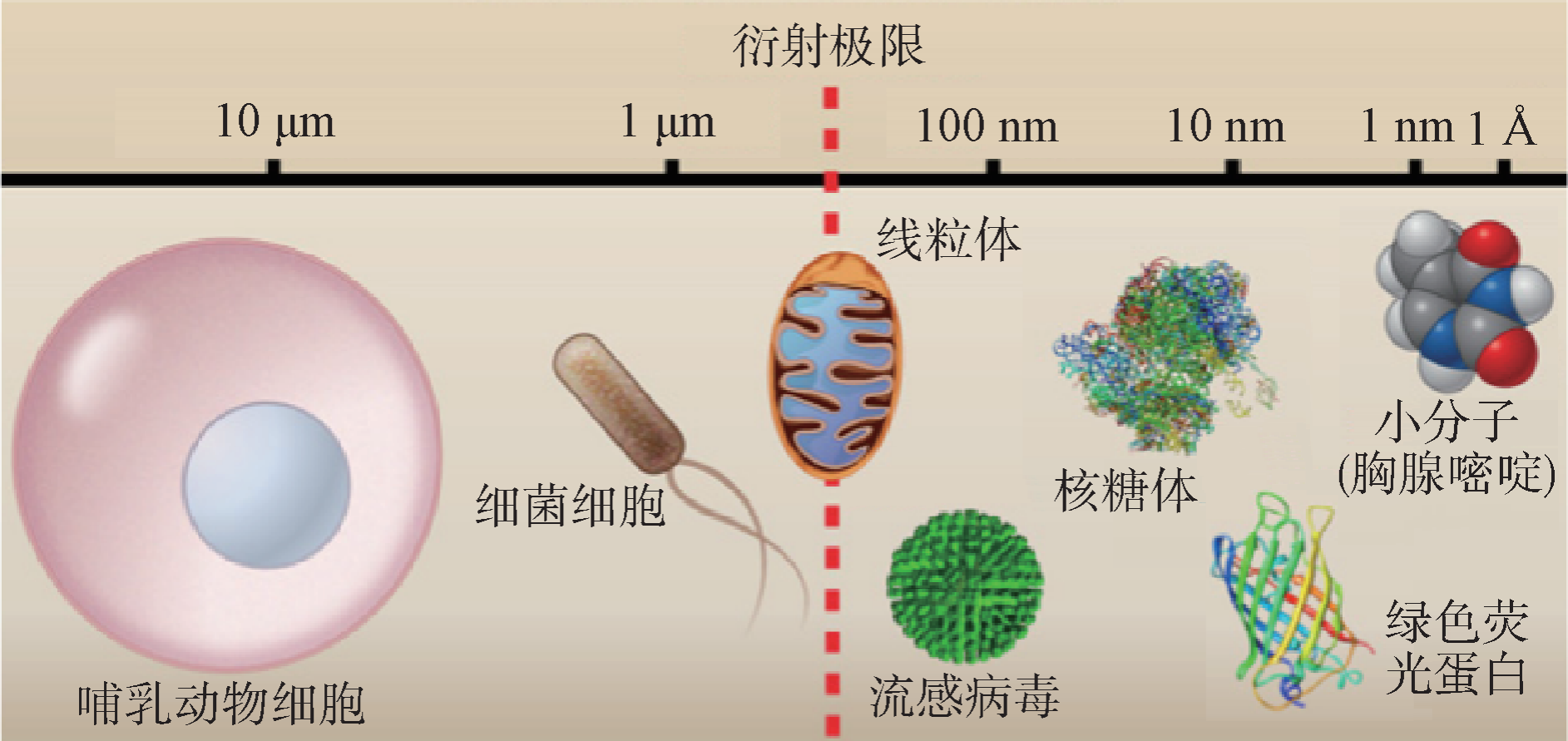
图1 不同生物样品尺寸与衍射极限比较[1]
深入研究细胞的生命活动,需要提高显微镜的分辨率和成像衬度这两个关键指标。要观察细胞内的精细结构,最重要的是分辨率。分辨率是指能区分的两个点之间的最小距离(图2)。一个理想的点光源在通过显微镜的透镜系统后,其光束会聚集成一个微小的光斑。在这个过程中,由于光的衍射现象,光斑的大小会随着波长和显微镜数值孔径而变化。当两个光斑靠得太近时,他们之间的界限变得模糊不清,从而无法分辨出它们各自的位置。这种现象导致了显微镜存在一个分辨率极限。换句话说,如果两个点光源的距离小于分辨率极限,我们就无法分辨出两个物体。因此,分辨率极限是光学显微镜的一个重要限制因素,它限制了显微镜能够分辨出的最小物体的尺寸。

图2 衍射极限的概念(图片来自维基百科)
19世纪,以德国物理学家阿贝为代表的研究者通过各种方式大幅改善了显微镜的分辨率,如利用油镜来增加物镜的数值孔径,采用更好的像差校正技术来设计显微镜的光学系统等。然而阿贝也于1873年提出,显微镜的分辨率受到光学衍射极限的限制[2],横向和轴向分辨率理论极限分别由下式所决定:
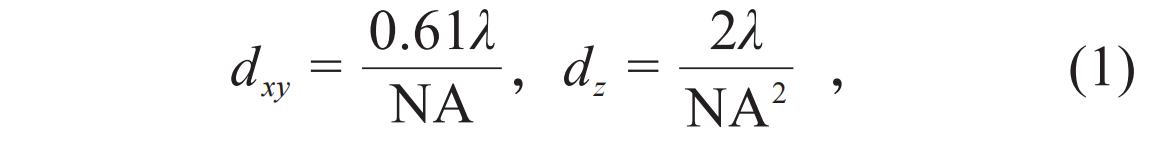
其中,dxy和dz分别表示传统光学显微镜的横向、轴向分辨率,λ表示光的波长,NA表示所使用物镜的数值孔径。由此可见,在可见光波段(400—700 nm),传统光学显微镜的横向分辨率只能达到200—300 nm,轴向分辨率极限在500—800 nm。也就是说,如果两个目标之间的距离小于两三百纳米,我们便无法区分它们。由于很多亚细胞结构如细胞骨架、细胞器、细胞内膜结构等都在数纳米到数百纳米的尺度,因此用传统显微镜难以对其进行研究。
除了分辨率,衬度也是显微镜成像重要的指标。细胞对可见光有较高的透过率,以更高的对比度观察细胞是细胞生物学研究中亟待解决的问题。为了解决这个问题,科学家们采用了许多技术手段来增强显微镜下细胞的对比度。20世纪相衬显微镜和微分干涉显微镜的提出,为生物学和医学领域带来了重大的突破,使得我们能够观察到透明细胞的细节[3,4]。这两种成像技术通过将细胞内微小的折射率变化转换成可见的亮度变化,从而使观察透明细胞的微小结构成为可能。
另一项革命性的技术是荧光标记以及荧光显微镜技术。这种技术利用特殊的荧光染料或荧光蛋白标记细胞中的特定结构或蛋白质,从而使其在显微镜下更容易被发现。随着荧光标记技术在20世纪被发明,将细胞特定结构标记上荧光探针,就能清晰地观察各种亚细胞结构。与传统的染色技术不同,荧光标记具有高灵敏度、高特异性和高分辨率等优点,可以清晰地观察各种亚细胞结构,包括细胞器、细胞膜、染色体等。荧光成像具有非常高的对比度,配合高灵敏光学检测系统,甚至可以实现单个荧光分子信号的检测。通过将特定蛋白连接荧光分子,可以在细胞内对单个蛋白进行定位和追踪,这在之前是无法实现的。荧光显微镜为研究亚细胞结构和生物大分子功能供了强有力的工具。
荧光标记技术也推动了显微镜技术的发展,20世纪50年代发明的共聚焦显微镜(confocal laser scanning microscope,CLSM)[5],通过将光束汇聚成一个小点来扫描样品,并通过一个针孔过滤背景荧光,从而实现光学层析成像。它可以利用点扫描技术获得细胞或组织三维图像,提高图像的清晰度和分辨率,克服了普通荧光显微镜成像模糊、三维分辨率低的缺点。它的发明标志着光学显微成像技术进入了高清三维成像时代。
STED的概念最早由德国马普所的Stefan W. Hell教授于1994年提出,它采用点扫描的方式成像,将激发光(图3(a)绿色光路)和中心光强为零的环形损耗光(红色光路)同时聚焦在样品上[6]。由于荧光分子受激发射产生的信号波长与损耗光相同,可以被滤光片滤掉,因此探测器接收到的被绿光激发的荧光信号(黄色光路)就是损耗光中心光强为零的区域的信号。使用这种方法,可以将等效的成像区域尺寸缩小到衍射极限以下,实现超分辨成像(图3(b))。STED的分辨率可以到数十纳米[7—9]。
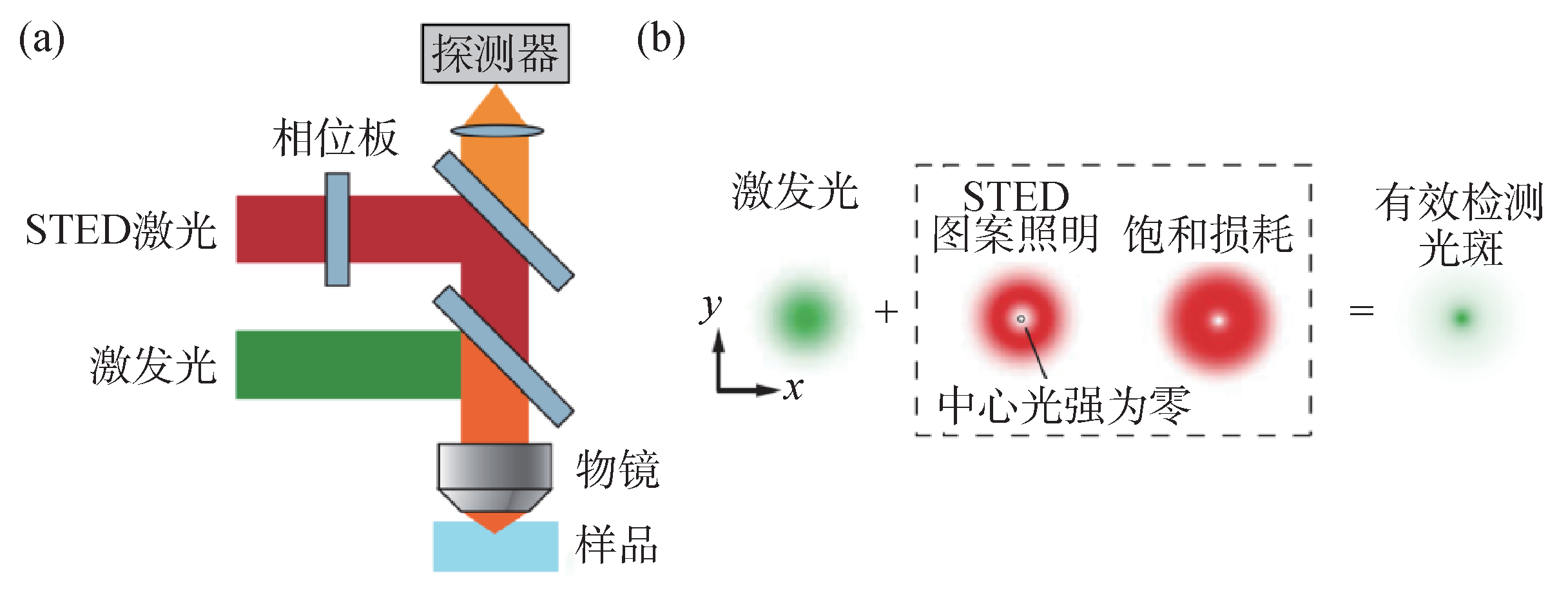
图3 (a)STED技术所用成像光路;(b)STED原理图[6]
SIM是一种基于面照明和相机检测的成像技术,通过将目标的高频信息移到低频部分上,拓宽了可以被显微镜系统捕获的空间频率范围,进而实现样品结构细节的解析。在光学显微成像中,由于物镜的收集能力限制,样品的高频信息会被丢失,无法被显微镜捕获和记录。SIM技术通过莫尔条纹的叠加,可以将这些位于物镜收集能力范围之外的高频信息“搬运”到低频区域,从而让显微镜能够捕获到这些原本无法获取的信息(图4)。

图4 SIM技术原理[10]。未知样品的高频信息表现为细条纹,当未知样品与已知结构光照明图案相重叠,未知样品的高频信息被移到低频处,表现为较宽的条纹
在采集结构光照明的样品图像后,SIM技术采用特殊的图像算法处理这种叠加的低频信息和高频信息,从而获取超分辨图像。这种超分辨图像的分辨率能够比普通光学显微镜提高一倍左右[10,11]。SIM技术的优势是能够在极低光强下实现超分辨率成像,它在时间分辨率以及低光毒性等方面有独特的优势。因此非常适合针对活细胞动态过程的长时间观察。
SMLM的技术原理是,对一个单分子的位置测量的误差可以远远小于其光斑的尺寸,因此如果可以对样品中所有荧光分子的位置进行重构,就可以突破衍射限制,实现对细胞内纳米结构更高的分辨能力。
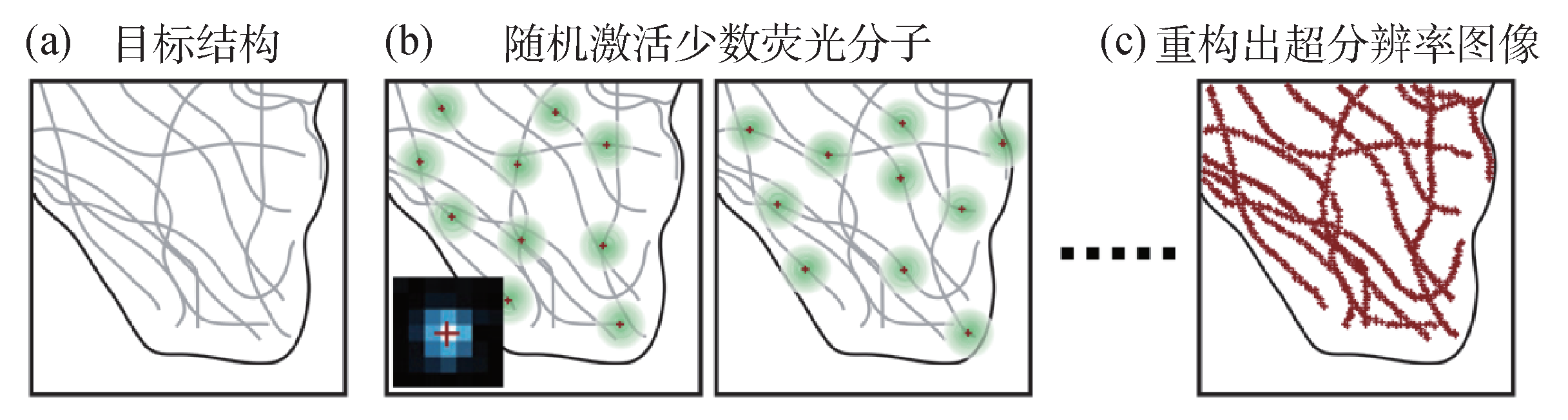
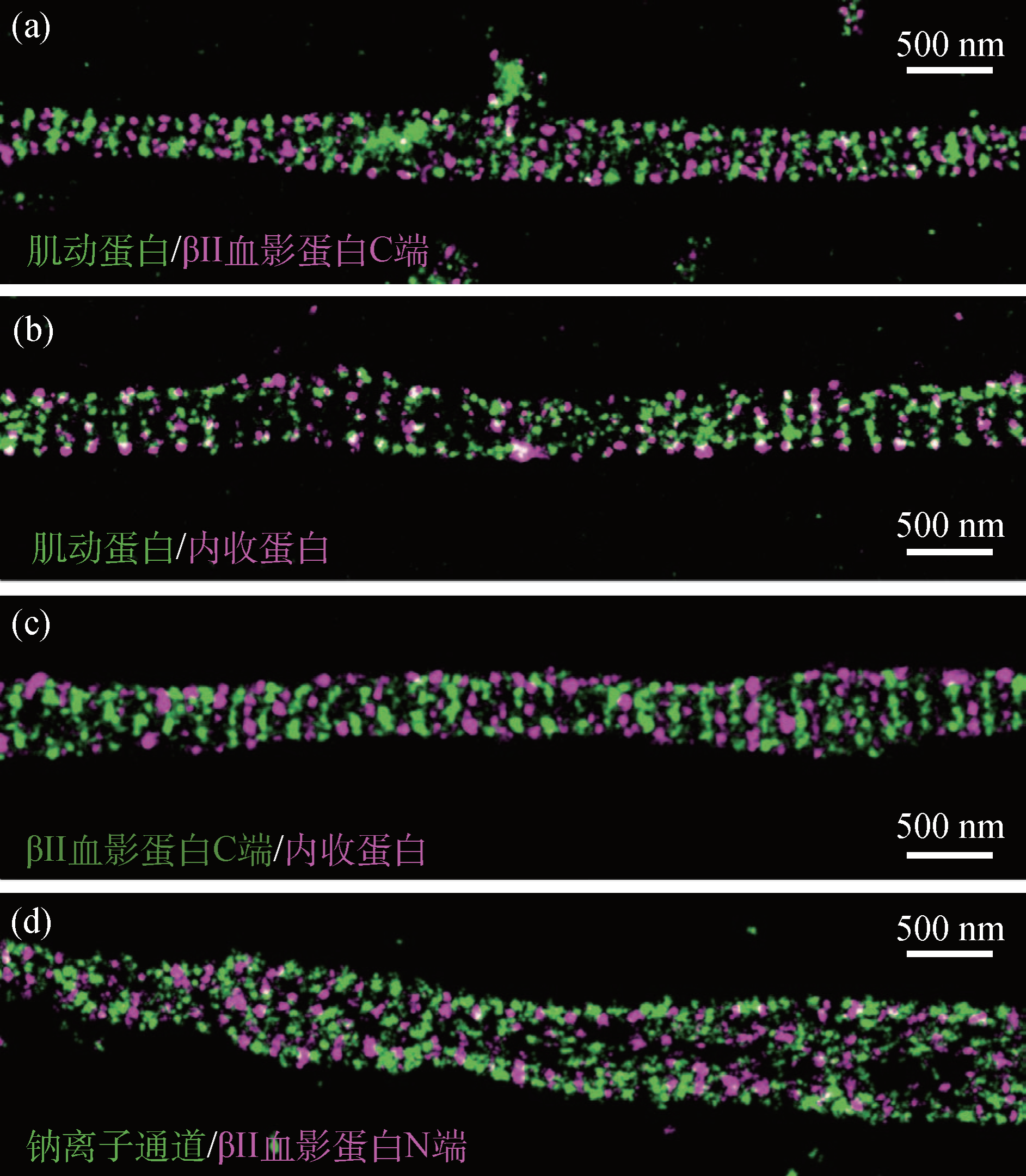
图6 SMLM技术解析神经轴突周期性结构[14]。分别对神经元轴突中肌动蛋白(图(a),(b)中的绿色通道),βII血影蛋白(图(a),(d)中的红色通道,图(c)中的绿色通道),内收蛋白(图(b),(c)中的红色通道)以及钠离子通道(图(d)中的绿色通道)的分布进行超分辨率解析,发现这些组分呈现周期性交叉排列。此前使用传统显微成像技术一直无法观察到这种结构。这一结构的解析对于轴突的形态和功能研究具有重要意义
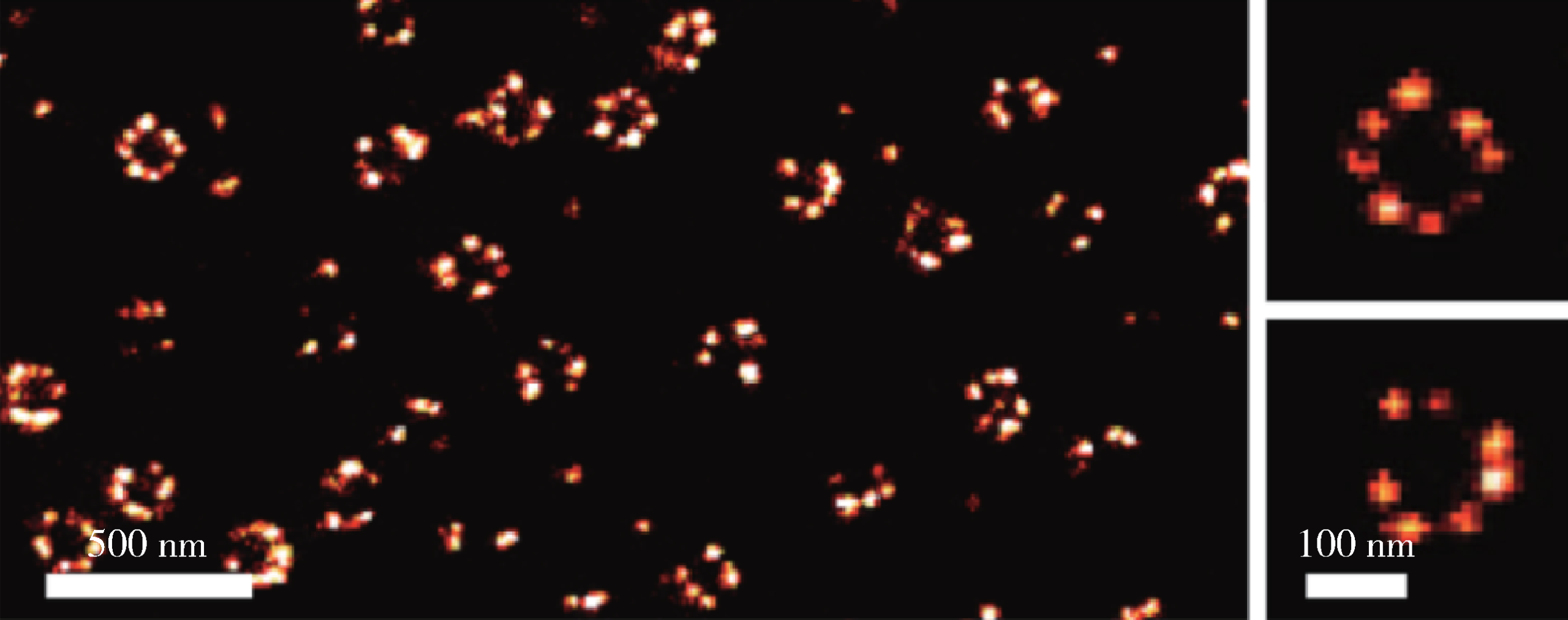
图7 SMLM技术解析核孔复合物[15]。左图为NPC成像结果,右侧两图为典型的NPC八聚体结构。通过解析核孔复合物的八聚体结构,说明SMLM技术在解析生物大分子复合物方面具有很大潜力
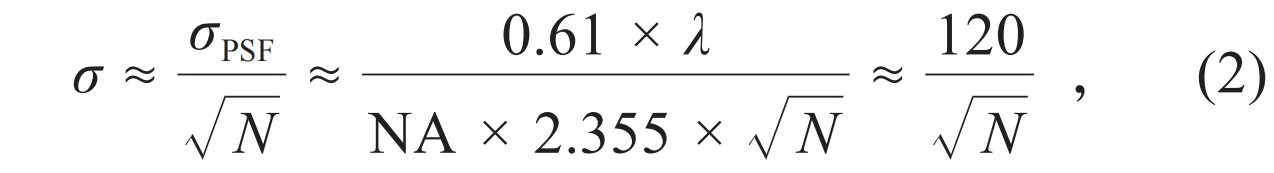
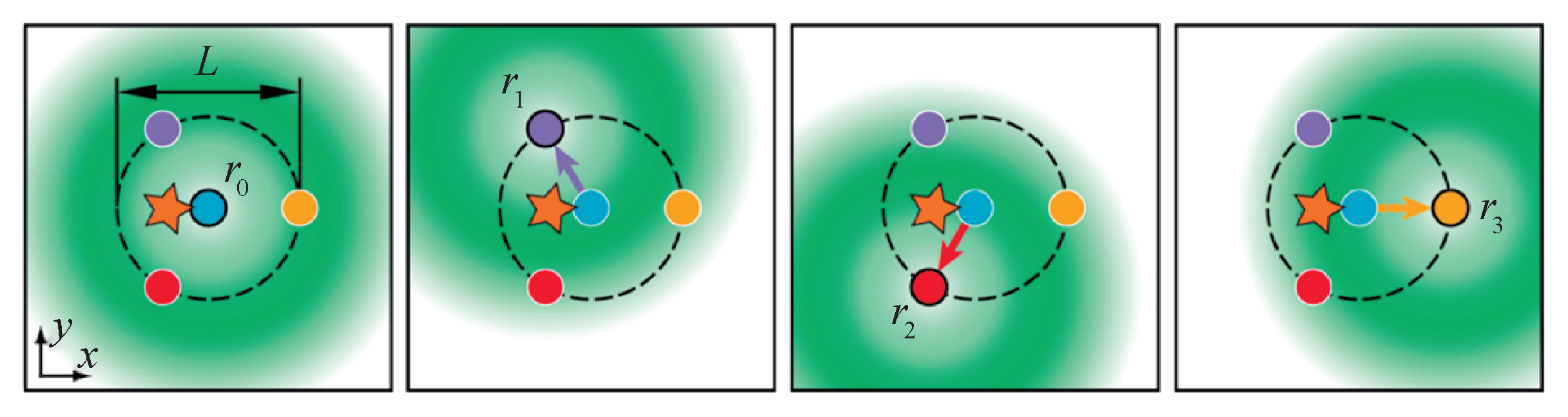
图8 MINFLUX原理图[15,16]。图中黄色五角星表示荧光分子,四个颜色圆点表示四次照明的光斑中心位置,绿色部分表示实际的空心照明光斑。通过使用空心照明光斑围绕单分子进行四次激发,实现对单分子的精确定位
MINFLUX技术虽然具有非常高的空间分辨率,但也存在一个缺点,即该技术利用单个甜甜圈状的光点激发荧光分子,因此同一时间只能定位或追踪一个分子,成像通量太低,数据收集所需时间较长。特别是对于较大或较致密的细胞结构来说,不适合应用该技术解析。
为了在不损失成像速度和通量的前提下提升单分子定位成像的分辨率,中国科学院生物物理研究所的团队发展了干涉单分子定位显微镜技术[17,18]。同样是利用面照明和相机高通量检测,与传统质心拟合定位方法不同的是,这项技术采用了干涉定位代替现有的单分子图像质心定位,通过利用激光干涉产生明暗相间的条纹来激发荧光分子,进一步提高了单分子的定位精度。这种单分子定位方法被命名为重复光学选择性曝光技术(ROSE)[17],其定位精度公式如下:
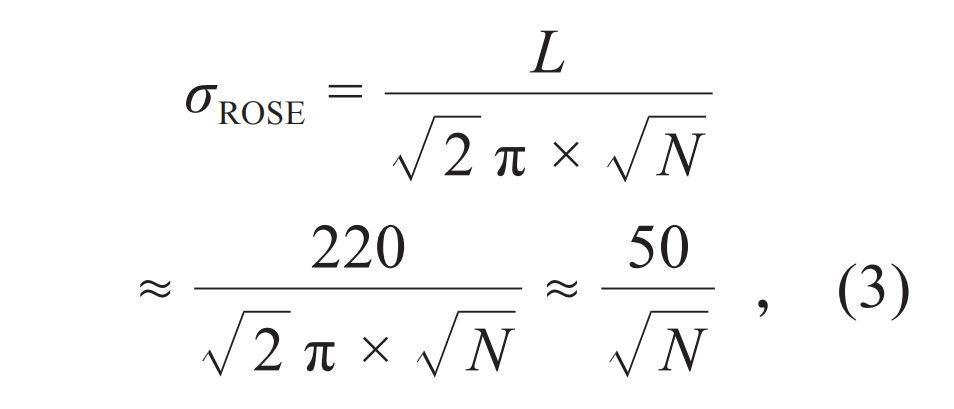
式中L是干涉条纹的周期,取决于波长和物镜的有效数值孔径,实测中能达到220 nm。在相同光子数条件下,ROSE的理论定位精度能够达到质心拟合的2.4倍。
ROSE定位原理与卫星定位类似。在卫星定位中,通过测量几颗卫星与目标的距离,即可确定目标在地球上位置。在ROSE技术中采用的干涉条纹就起到卫星的作用,3种相位间隔120°的干涉条纹被用来激发荧光分子,条纹的周期和相位都是已知的,不同条纹激发出不同强度的荧光信号,可用来确定目标分子在条纹里的相对位置。基于这个定位原理,单分子定位成像的分辨率被进一步提升到纳米级别,研制的纳米分辨率显微镜可以对细胞内微小结构的细节进行观察和分析,满足生物学研究对分辨率越来越高的需求。
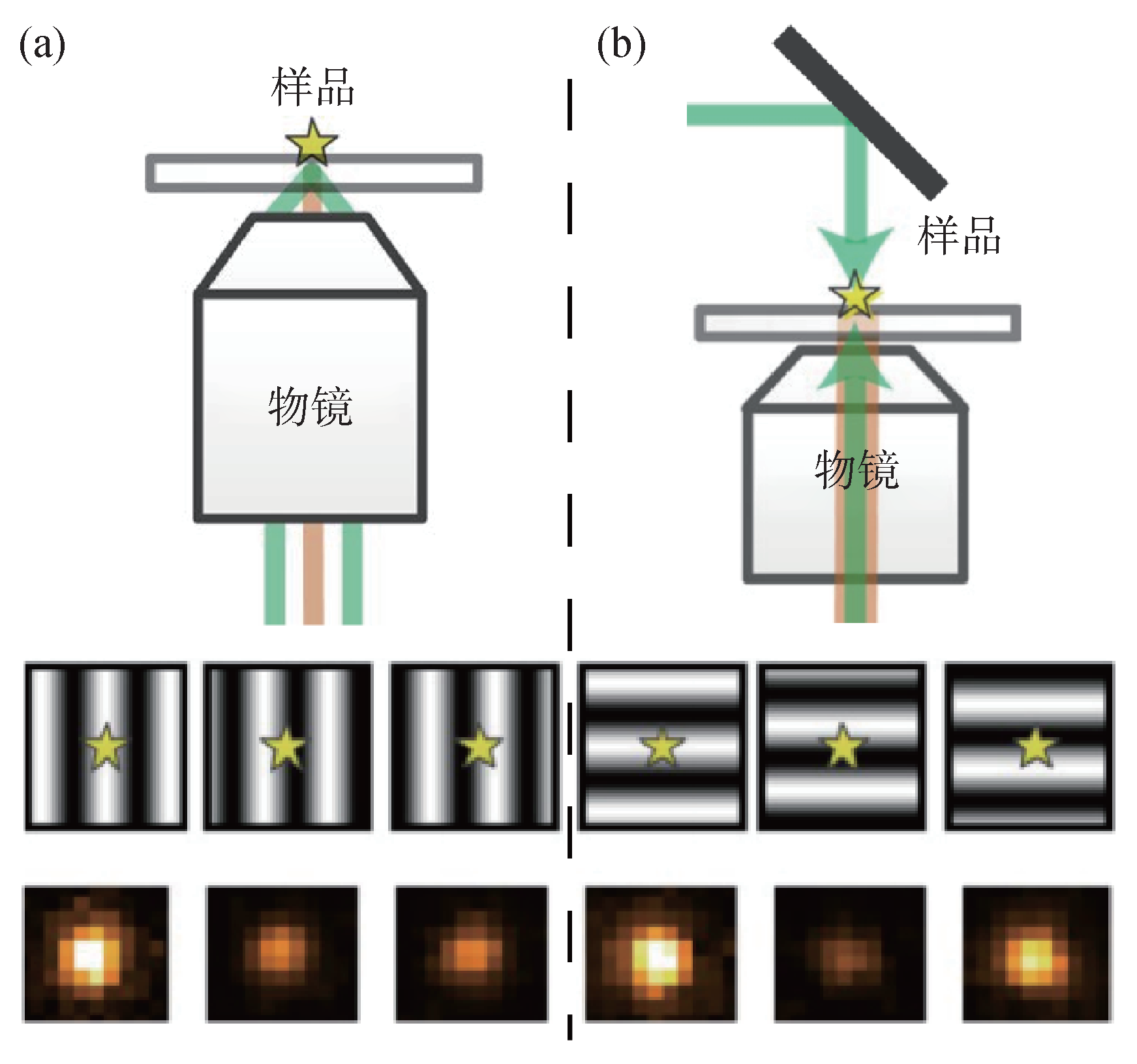
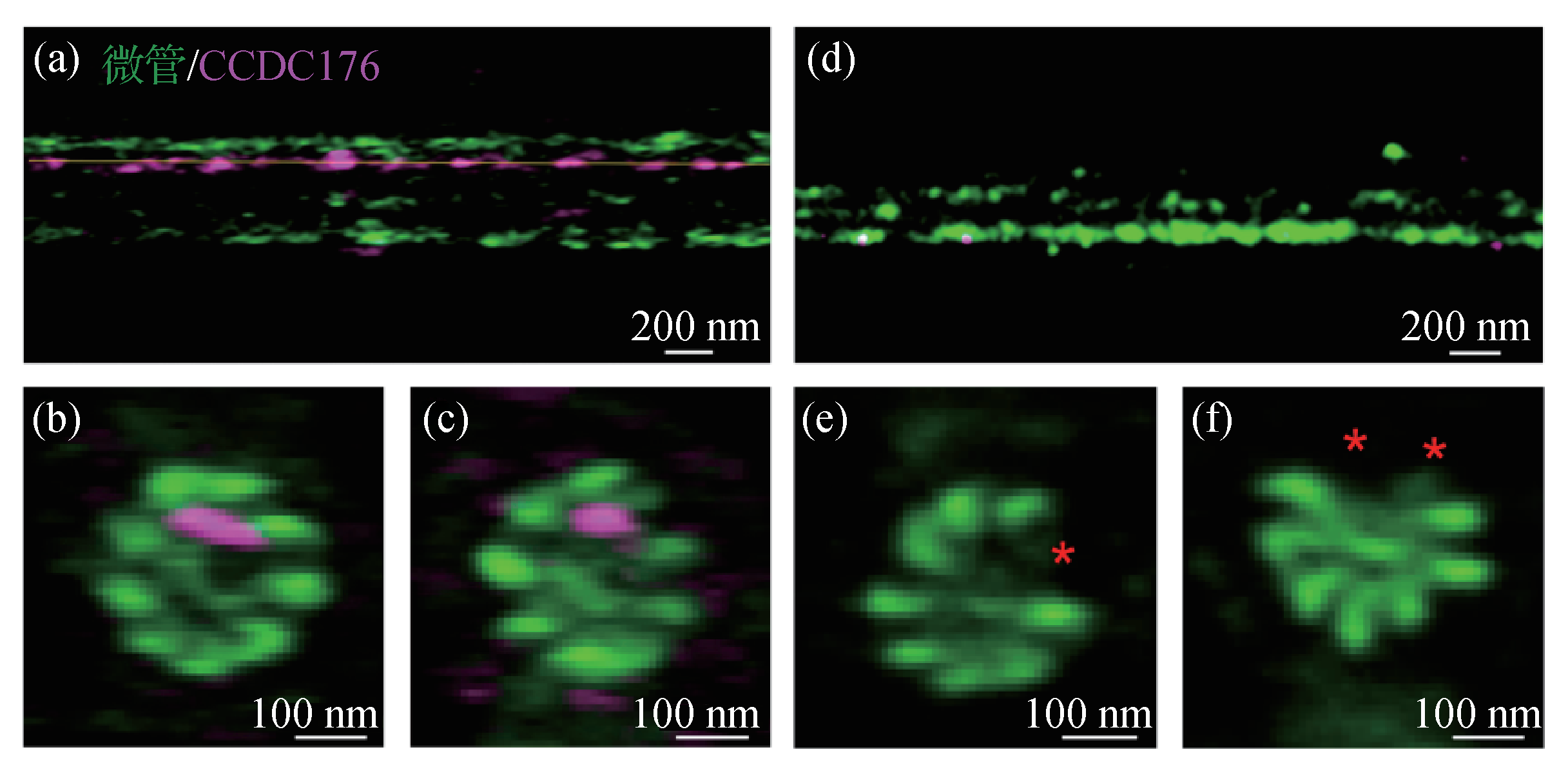
图10 ROSE-Z成像正常精子鞭毛轴丝纵截面(a)和横截面(b)、(c),及CCDC176敲除的精子鞭毛纵截面(d)和横截面(e)、(f)[23]。图中微管为绿色通道,在横截面图(b),(c)中双联微管围绕中心呈环状结构,敲除CCDC176后存在部分缺失(图(e),(f),星号表示缺失部位)。CCDC176为红色通道,位于双联管内侧非对称分布(图(a)—(c))
超分辨成像技术突破了传统光学显微镜衍射极限的限制,实现了对细胞内各种精细结构的观察以及目标蛋白分子定位分布的研究。以单分子定位显微镜技术为代表的多种超分辨显微成像技术的不断发展,为揭示生命的奥秘提供了一种强有力的研究工具。
近几年发展的纳米分辨率单分子定位成像技术,与传统基于单分子图像拟合的质心定位技术不同,利用具有不同强度空间分布的光场激发荧光分子,通过分析单个荧光分子的信号强度及与光场关系来判断分子的空间位置。这一新技术的出现为生命科学研究提供了新的手段,并逐渐应用于生物大分子机器原位成像研究。如近期Ardem Patapoutian课题组使用纳米分辨率成像技术,结合定点插入非天然氨基酸和点击化学标记技术,对机械力敏感离子通道Piezo 1蛋白在细胞膜上进行原位成像。证明了该蛋白亚基末端具有高度灵活性,也是电镜无法解析该区域结构的原因,并通过成像小分子激动剂、抑制剂下的蛋白末端,获取距离信息,解释了Piezo 1蛋白构象变化的机理[24]。
单分子定位超分辨技术在光学成像领域拥有最高的空间分辨率,与光片和自适应光学等技术结合,可以实现全细胞超分辨成像;而将ROSE-XY与ROSE-Z结合的ROSE-3D技术,有望可以使不同深度的荧光分子定位精度保持在同样高的水平,实现厚样品的各向同性超分辨成像。随着越来越多的单分子成像和分析方法的出现,再结合深度学习等技术,超分辨荧光显微镜在更高时空分辨率、深层成像、活细胞成像等各方面的提升优化,将不断推动生命科学、医学等研究领域的进步。
[2] Abbe E. Archiv für Mikroskopische Anatomie,1873,9(1):413 [3] Zernike F. Physica,1942,9(10):974 [4] Nomarski G. J. Phys. Radium,1955,16:9S [5] Minsky M. Scanning,1988,10(4):128 [6] Huang B,Bates M,Zhuang X. Annu. Rev. Biochem.,2009,78:993 [7] Hell S W,Kroug M. Applied Physics B,1995,60(5):495 [8] Hell S W,Wichmann J. Optics Letters,1994,19(11):780 [9] Klar T A et al. Proceedings of the National Academy of Sciences,2000,97(15):8206 [10] Gustafsson M G L. Proceedings of the National Academy of Sciences,2005,102(37):13081 [11] Gustafsson M G L. Journal of Microscopy,2000,198(2):82 [12] Betzig E et al. Science,2006,313(5793):1642 [13] Rust M J,Bates M,Zhuang X. Nature Methods,2006,3(10):793 [14] Xu K,Zhong G,Zhuang X. Science,2013,339(6118):452 [15] Szymborska A et al. Science,2013,341(6146):655 [16] Balzarotti F et al. Science,2017,355(6325):606 [17] Gu L et al. Nature Methods,2019,16(11):1114 [18] Gu L et al. Nature Methods,2021,18(4):369 [19] Thompson R E,Larson D R,Webb W W. Biophysical Journal,2002,82(5):2775 [20] Huang B et al. Science,2008,319(5864):810 [21] Juette M F et al. Nature Methods,2008,5(6):527 [22] Pavani S R P et al. Proceedings of the National Academy of Sciences,2009,106(9):2995 [23] Liu C et al. Current Biology,2023,33:3371 [24] Mulhall E M et al. Nature,2023,620:1117
免责声明:本文旨在传递更多科研资讯及分享,所有其他媒、网来源均注明出处,如涉及版权问题,请作者第一时间联系我们,我们将协调进行处理,最终解释权归旭为光电所有。